怎样用java 做网站营业推广方案怎么写
🌞欢迎来到图神经网络的世界
🌈博客主页:卿云阁💌欢迎关注🎉点赞👍收藏⭐️留言📝
🌟本文由卿云阁原创!
📆首发时间:🌹2024年3月20日🌹
✉️希望可以和大家一起完成进阶之路!
🙏作者水平很有限,如果发现错误,请留言轰炸哦!万分感谢!
目录
GNN起源
图的矩阵表示
层内与层间的消息传递
GCN
GraphSAGE
代码实战
GAT
代码实战
GNN起源
(1)数学中的空间有很多种,大部分都是定义在欧氏里德空间的,比如图像,文本。除此之外还存在着大量的非欧空间,比如分子结构。
(2) 图嵌入常⻅模型有DeepWalk,Node2Vec等,然而,这些方法方法有两种严重的缺点,首先就是节点编码中权重未共享,导致权重数量随着节点增多而线性增大,另外就是直接嵌入方法缺乏泛化能力,意味着无法处理动态图以及泛化到新的图。
如何把这种图结构嫁接到神经网络上,图神经网络就诞生了。和传统的神经网络结构相比,它解决了两个问题。
- 图结构的矩阵画表示
- 层内与层间的消息传递
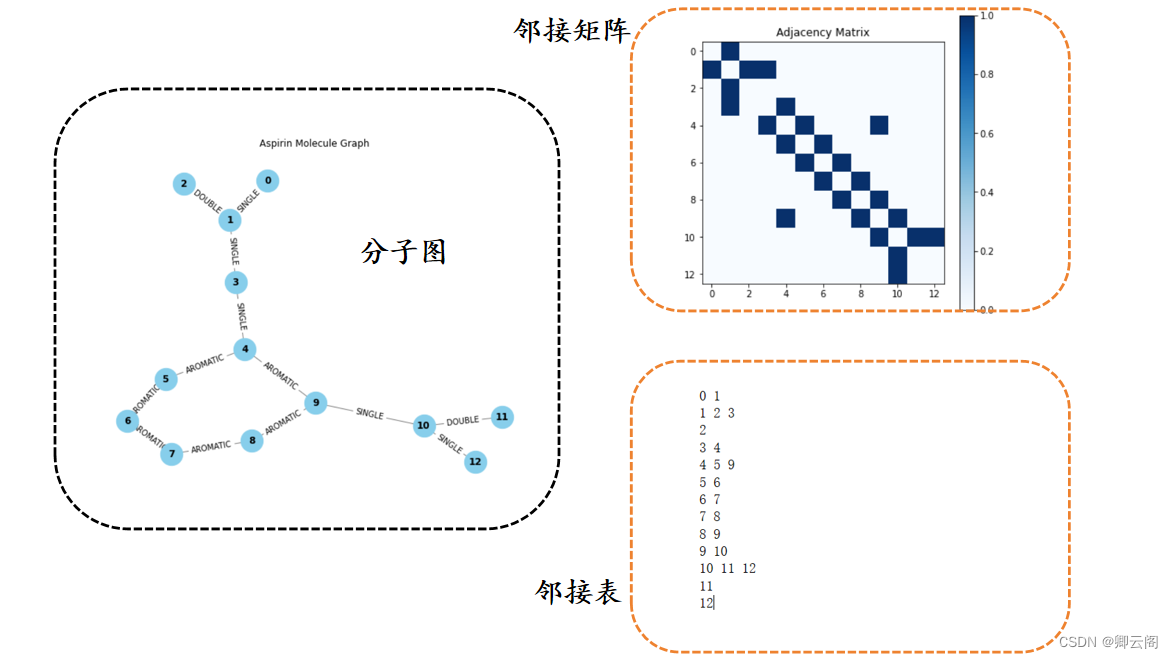
图的矩阵表示
- 借用邻接矩阵
- 考虑稀疏性,还可以使用邻接表。
层内与层间的消息传递
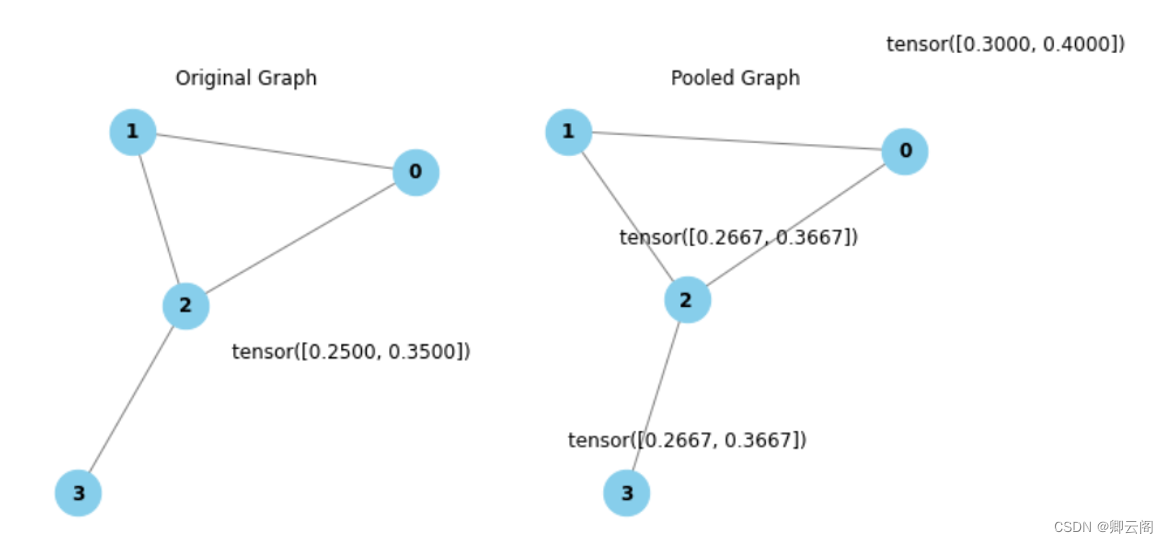
聚合
简单来说一个节点或者边的特征,不光看它自己,还要由它相邻元素的加权求和决定。层内的聚合常常被称之为池化。
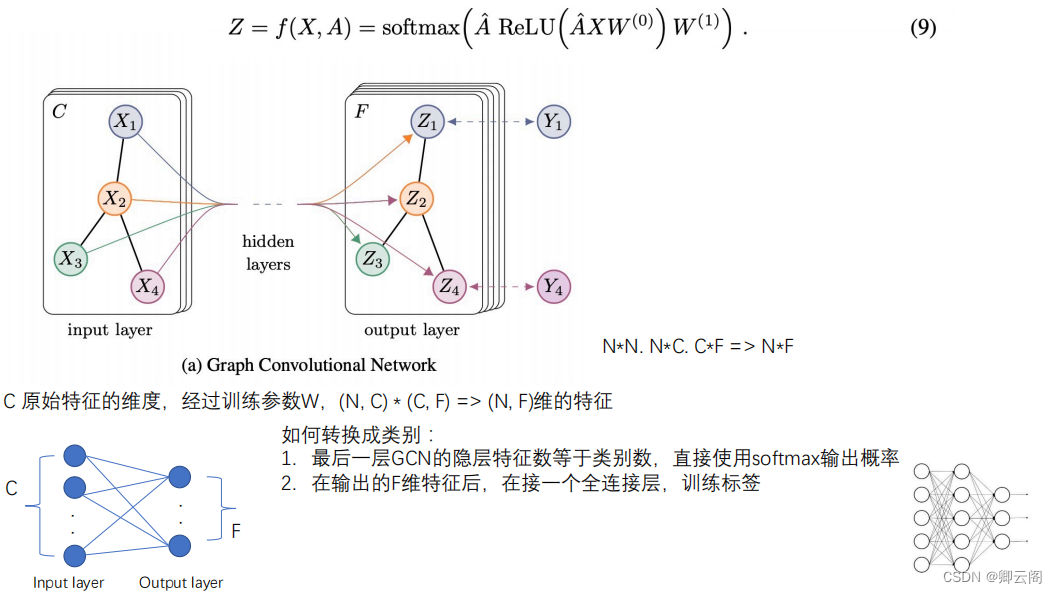
层级间的关系传递,通过节点的连接关系进行,也可以看成是一种聚合,根据聚合方法的差异形成了不同的算法,最简单的是图卷积网络GCN。就是在层间经过邻域聚合实现卷积特征提取。左乘于邻接矩阵表示对每个节点来说,该节点的特征为邻域节点的特征,相加之后的结果。
如果聚合的时候没有用全部的邻域节点,而是先采样再聚合,就是GraphSAGE算法。
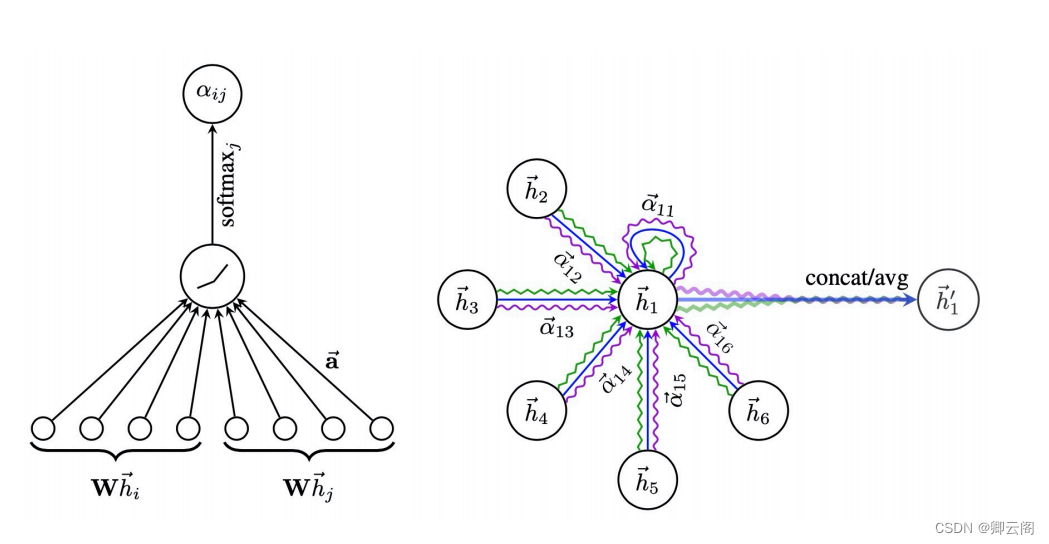
如果聚合的时候考虑了领域节点的权重,也就是运用了注意力机制,那么就是图注意力网络GAT。
聚合还可以用在非监督模型上,比如把图和变自分编码器相结合,形成GAE算法。
除此之外还有更复杂的图生成网络,和图时空网络。
GCN
原理解析:
代码实战:
import torch import torch.nn as nn import dgl import dgl.function as fn import networkx as nx import matplotlib.pyplot as plt from rdkit import Chem from rdkit.Chem import Draw# 构建阿司匹林分子 aspirin_smiles = "CC(=O)OC1=CC=CC=C1C(=O)O" aspirin_mol = Chem.MolFromSmiles(aspirin_smiles)# 构建分子图 aspirin_graph = dgl.from_networkx(nx.Graph(Chem.rdmolops.GetAdjacencyMatrix(aspirin_mol)))# 可视化分子结构 Draw.MolToImage(aspirin_mol)# 定义GCN模型 class GCN(nn.Module):def __init__(self, in_feats, hidden_size, num_classes):super(GCN, self).__init__()self.conv1 = dgl.nn.GraphConv(in_feats, hidden_size)self.conv2 = dgl.nn.GraphConv(hidden_size, num_classes)def forward(self, g, features):h = self.conv1(g, features)h = torch.relu(h)h = self.conv2(g, h)return h# 初始化GCN模型 input_dim = 1 # 输入特征维度为1,因为我们只考虑一个原子的属性 hidden_size = 64 num_classes = 2 # 为简单起见,假设我们的任务是二分类 gcn_model = GCN(input_dim, hidden_size, num_classes)# 可视化GCN模型结构 print(gcn_model)# 可视化分子图 plt.figure(figsize=(8, 6)) nx.draw(aspirin_graph.to_networkx(), with_labels=True, node_color='skyblue', node_size=800, font_size=12, font_weight='bold', edge_color='gray') plt.title('Molecular Graph') plt.show()
GraphSAGE
代码实战
我们来实现了一个简单的 GraphSAGE 模型,并对阿司匹林的分子结构进行预测。首先,我们需要构建一个简单的图结构来表示阿司匹林的分子。然后,我们将定义一个GraphSAGE 模型,并使用该模型对阿司匹林分子的属性进行预测。
import torch import torch.nn as nn import dgl import dgl.function as fn import networkx as nx import matplotlib.pyplot as plt import numpy as np# 构建一个简单的分子图来表示阿司匹林的结构 aspirin_graph = dgl.graph(([0, 1, 1, 2], [1, 0, 2, 1])) # 定义边的连接关系# 可视化分子图 plt.figure(figsize=(4, 4)) nx.draw(aspirin_graph.to_networkx(), with_labels=True, node_color='skyblue', node_size=800, font_size=12, font_weight='bold', edge_color='gray') plt.title('Molecular Graph') plt.show()# 定义GraphSAGE模型 class GraphSAGE(nn.Module):def __init__(self, in_feats, hidden_size, num_classes):super(GraphSAGE, self).__init__()self.conv1 = dgl.nn.SAGEConv(in_feats, hidden_size, 'mean')self.conv2 = dgl.nn.SAGEConv(hidden_size, num_classes, 'mean')def forward(self, g, features):h = self.conv1(g, features)h = torch.relu(h)h = self.conv2(g, h)return h# 初始化GraphSAGE模型 input_dim = 1 # 输入特征维度为1,因为我们只考虑一个原子的属性 hidden_size = 64 num_classes = 2 # 为简单起见,假设我们的任务是二分类 graphsage_model = GraphSAGE(input_dim, hidden_size, num_classes)# 生成随机的示例数据 num_samples = aspirin_graph.number_of_nodes() node_features = torch.randn(num_samples, input_dim)# 随机生成二分类标签(示例) labels = torch.randint(0, 2, (num_samples,))# 将标签添加到图中的节点 aspirin_graph.ndata['features'] = node_features aspirin_graph.ndata['labels'] = labels# 定义损失函数 loss_fn = nn.CrossEntropyLoss()# 模型训练 optimizer = torch.optim.Adam(graphsage_model.parameters(), lr=0.001) epochs = 50for epoch in range(epochs):logits = graphsage_model(aspirin_graph, aspirin_graph.ndata['features'])loss = loss_fn(logits, aspirin_graph.ndata['labels'])optimizer.zero_grad()loss.backward()optimizer.step()if (epoch + 1) % 10 == 0:print(f'Epoch [{epoch+1}/{epochs}], Loss: {loss.item()}')# 使用模型进行预测(示例) with torch.no_grad():predicted_labels = torch.argmax(graphsage_model(aspirin_graph, aspirin_graph.ndata['features']), dim=1)print("Predicted Labels:", predicted_labels)
GAT
代码实战
import torch import torch.nn as nn import dgl import dgl.function as fn import networkx as nx import matplotlib.pyplot as plt# 构建阿司匹林分子的简单图结构 aspirin_graph = dgl.graph(([0, 0, 0, 1, 2], [1, 2, 3, 3, 3])) # 使用边列表构建图# 定义节点特征 node_features = torch.tensor([[0.1, 0.2],[0.2, 0.3],[0.3, 0.4],[0.4, 0.5] ], dtype=torch.float)# 将节点特征设置到图中 aspirin_graph.ndata['feat'] = node_features# 可视化分子图 plt.figure(figsize=(8, 6)) nx.draw(aspirin_graph.to_networkx(), with_labels=True, node_color='skyblue', node_size=800, font_size=12, font_weight='bold', edge_color='gray') plt.title('Molecular Graph') plt.show()class GAT(nn.Module):def __init__(self, in_dim, hidden_dim, out_dim, num_heads):super(GAT, self).__init__()self.conv1 = dgl.nn.GATConv(in_dim, hidden_dim, num_heads)self.conv2 = dgl.nn.GATConv(hidden_dim * num_heads, out_dim, num_heads)def forward(self, g, features):h = self.conv1(g, features)h = torch.relu(h)h = self.conv2(g, h)return h# 初始化 GAT 模型 input_dim = 2 # 输入特征维度 hidden_dim = 64 out_dim = 1 # 输出维度,这里假设我们只需要一个输出维度进行二分类 num_heads = 2 gat_model = GAT(input_dim, hidden_dim, out_dim, num_heads)# 输出 GAT 模型结构 print(gat_model)